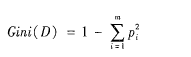随机森林介绍
随机森林是一种用于解决回归或者分类问题的算法,它是组合分类的一个应用,组合分类就是把K个学习得到的模型M1,M2,MK全部组合在一起,创建一个改进的复合分类模型M,组合分类器基于分类器的投票返回类预测;组合分类器往往比它的基分类更准确,组合分类中还有想装袋,提升等;
随机森林的估计过程
A、指定M值,即随机产生M个属性用于节点上的二叉树,二叉树属性选择任然满足不纯度最小原则,不纯度公式为
B、应用BOOTSTRAP自助法在员数据集中有放回地随机抽取K个样本集,组成K颗决策树,而对于未被抽取的样本用于决策树的预测;
C、根据K个决策树组成的随机森林对待分类样本进行分类或者预测,分类的原则是投票法,预测的原则是简单平均。
想象组合分类器中每个分类器都是一颗决策树,因此分类器的集合就是一个“森林”,使用CART算法的方法来增长树,树增长到最大的规模,并且不剪枝,用这种方式形成的随机森林称为Forest-RI,
另一种形式称为Forest-RC,他不是随机地选择一个属性子集,而是选择一个属性子集,而是由已有的属性的线性组合创建一些新属性,就是由原来的S个属性组合,在给定的节点,随机选择S个属性,并且以次欧诺个[-1,1]中随机选取的数为系数相加,产生S个线性组合,并在其中找到最佳的划分,仅仅只有少量属性可用时,为了降低个体分类器
之间的相关性,这种形式的随机森林才有用。
模型评价因素
A`、根据树的生长,组成深林的分类性能就越好,因为树的增多森林的泛化误差收敛,那什么是泛化误差呢,泛化误差就是模型在真实情况(总体)上的表现出的误差就称为泛化误差,因此过拟合就不是我们考虑的问题;
B、每棵树之间的相关性越差,或者树之间是相互独立的,则森林的分类效果越好
R语言运用
R语言里面有个randomForest包的randomforest函数使用构建模型,importance()函数用于计算模型变量的重要性,MDSplot函数用于对随机森林的可视化,这里我使用莺尾花的数据集作为训练数据;
构建数据
构建测试和训练数据,70%作为训练数据,30%作为测试数据
library(randomForest)
sample_data <- sample(2,nrow(iris),replace=TRUE,prob=c(0.7,0.3))
sample_data
tra_data <- iris[sample_data==1,]
test_data <- iris[sample_data==2,]
选取合适的变量数
因为mtry参数是随机森林建模中,构造决策树分支时随机抽样的变量个数;选择合适的mtry值可以降低随机森林模型的预测误差;莺尾花数据总共有5个属性,因此写了遍历程序
R代码
for (i in 1:(m-1)){
test_model <- randomForest(Species~.,data=iris,mtry=i)
err <- mean(test_model$err)
print(err)
}
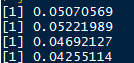
得到的结果如下,它告诉我mtry值在4的时候内平均误差是最低;因此确定参数mtry=4,建模,
选择合适的ntree值
ntree是指建模的时候决策树的数量,设置过低会导致预测误差过高,而ntree树过高会提升模型复杂度,降低效率;设随机种子为500,并查看结果,并可视化
tran_model <- randomForest(Species~.,data=iris,mtry=4,ntree=500)
plot(tran_model)
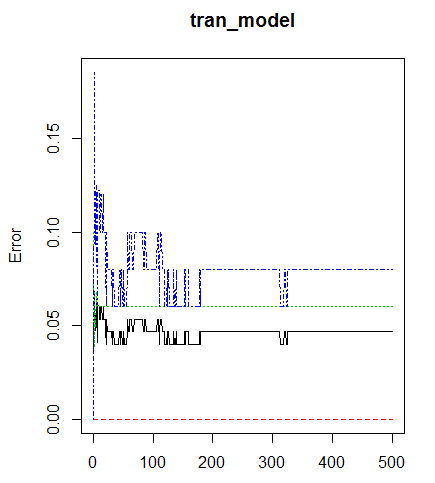
从上图看找出,当ntree=350的时候误差趋近于稳定,因此我们确定ntree=350;我们然后在建模
tran_model <- randomForest(Species~.,data=iris,mtry=4,ntree=350)
tran_model

从结果来看,模型误判率为4.67%,对分类为setosan误判为0,versicolor误判率为6%,对virginica误判8%;
变量重要性判断
我们这里使用Importance函数得到结果,并用varimpPlot()函数可视化
tran_imp <- importance(x=tran_model)
varImpPlot(tran_model)
tran_imp
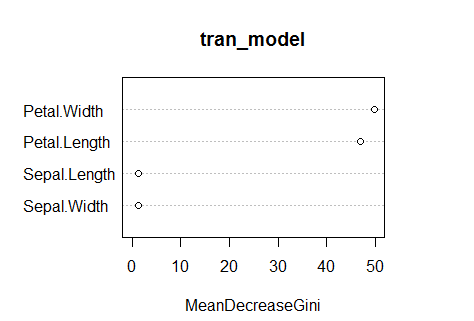
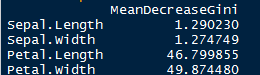
上面展示的各个变量的节点纯度,因此我可以这么认为,模型中的petal.length和petal.width最为重要
测试模型效果
table函数帮助我们判断,这里就不谢函数计算误差率了,有点懒
table(actual=test_data$Species,predicted=predict(tran_model,newdata = test_data,type = "class"))

so interesting!全对,看来模型很成功,就写到这里,有兴趣可以一起交流
参考文献
《数据挖掘:概念与技术(中文第三版)》
《R语言核心技术手册__第2版》