Part One:作业要求
数据源:PimaIndiansDiabetes2.csv
数据介绍:印第安人糖尿病数据库,包括以下信息:

作业需求:我们需要利用"pregnant","glucose", "pressure", "triceps", "insulin","mass",
"pedigree","age"等变量来预测"diabetes"的值。因为数据有缺失值,需要对数据先进行探索,完成缺失值的插补后,再利用分类算法建立预测模型,识别糖尿病患者。
项目要求:
1、 数据探索:利用课上讲到的列表显示缺失值和图形探究缺失数据两种方式对缺失值模式进行探究。
2、 数据完善:要求不能直接删除缺失数据,至少需要利用两种方式对缺失值进行插补。
3、 数据分区:需要按照变量diabetes来进行等比例抽样,其中80%作为训练集train数据,20%作为测试集test数据。
4、 建立模型及评估:利用分类算法对train数据集建立分类预测模型,并对test数据集进行预测,利用混淆矩阵查看模型评估效果。
Part Two:个人探索过程
1. 数据探索:
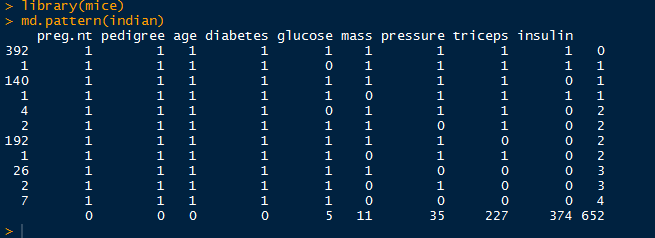
a. 列表显示缺失值
library(mice)
md.pattern(indian)
b. 图形探究缺失数据(aggr()函数)
library(VIM)
aggr(indian,prop=T,numbers=T)
aggr(indian,prop=F,numbers=T)
aggr(indian,prop=F,numbers=F)
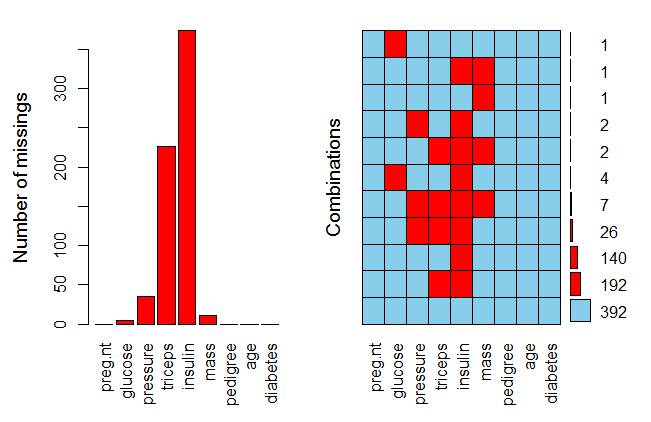
小结:根据列表和图形结果,共有五列数据存在缺失值:"glucose", "mass","pressure", "triceps", "insulin",分别缺失5,11,35,227,374个值。
2. 数据完善:
a. 随机森林插补
library(missForest) # 通过随机森林进行插补
z=missForest(indian)
indian.mf=z$ximp
md.pattern(indian.mf) # 列表验证
aggr(indian.mf,prop=T,numbers=T) # 图形验证
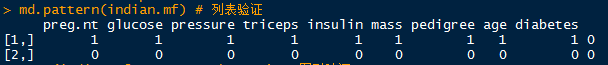
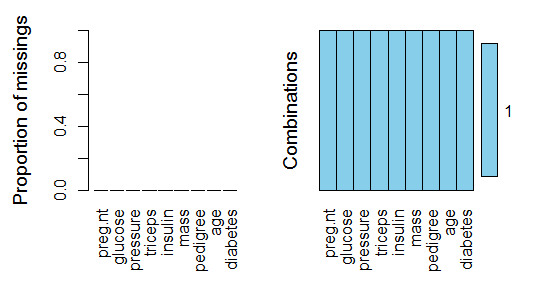
b. 回归模型插补
# 回归模型插补
indian.lm <- indian
# 插补glucose列缺失值
ind<-which(is.na(indian.lm[,2])==T) # 返回地2列为NA的行号
data_NL <- indian.lm[-ind,] #获取第2列所有不为NA的数据
data_NA <- indian.lm[ind,] #获取第2列为NA的数据
# 构建回归模型
lm = lm(glucose~preg.nt+pedigree+age,data=data_NL)
summary(lm) #查看模型的显著性:pedigree+age极其显著,preg.nt不显著
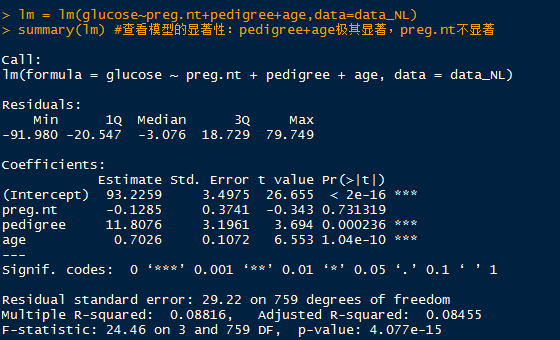
lm = lm(glucose~pedigree+age,data=data_NL) # 重新构建回归模型
indian.lm[ind,2] =round(predict(lm,data_NA)) #插补数据
依次通过以上方法对其它字段进行插补:
# 插补pressure列缺失值
ind<-which(is.na(indian.lm[,3])==T) # 返回地3列为NA的行号
data_NL <- indian.lm[-ind,] #获取第3列所有不为NA的数据
data_NA <- indian.lm[ind,] #获取第3列为NA的数据
# 构建回归模型
lm = lm(pressure~preg.nt+pedigree+age,data=data_NL)
summary(lm) #查看模型的显著性:age极其显著
lm = lm(pressure~age,data=data_NL) # 重新构建回归模型
indian.lm[ind,3] =round(predict(lm,data_NA))
# 插补triceps列缺失值
ind<-which(is.na(indian.lm[,4])==T) # 返回地4列为NA的行号
data_NL <- indian.lm[-ind,] #获取第4列所有不为NA的数据
data_NA <- indian.lm[ind,] #获取第4列为NA的数据
# 构建回归模型
lm = lm(triceps~preg.nt+pedigree+age,data=data_NL)
summary(lm) #查看模型的显著性:pedigree+age显著
lm = lm(triceps~pedigree+age,data=data_NL) # 重新构建回归模型
indian.lm[ind,4] =round(predict(lm,data_NA))
# 插补insulin列缺失值
ind<-which(is.na(indian.lm[,5])==T) # 返回地5列为NA的行号
data_NL <- indian.lm[-ind,] #获取第5列所有不为NA的数据
data_NA <- indian.lm[ind,] #获取第5列为NA的数据
# 构建回归模型
lm = lm(insulin~preg.nt+pedigree+age,data=data_NL)
summary(lm) #查看模型的显著性:preg.nt+pedigree+age显著
indian.lm[ind,5] =round(predict(lm,data_NA))
# 插补mass列缺失值
ind<-which(is.na(indian.lm[,6])==T) # 返回地6列为NA的行号
data_NL <- indian.lm[-ind,] #获取第6列所有不为NA的数据
data_NA <- indian.lm[ind,] #获取第6列为NA的数据
# 构建回归模型
lm = lm(mass~preg.nt+pedigree+age,data=data_NL)
summary(lm) #查看模型的显著性:pedigree显著
lm = lm(mass~pedigree,data=data_NL) # 重新构建回归模型
indian.lm[ind,6] =round(predict(lm,data_NA))
md.pattern(indian.lm) #列表验证 回归插补结果
aggr(indian.lm,prop=T,numbers=T) # 图形验证
小结:通过两种方式(随机森林,回归建模)对数据集进行插补得到两组数据集indian.mf、indian.lm。
3. 数据分区与建模
### 数据分区
library(caret)
# 构建result 存放结果
result <- data.frame(model=c("naiveBayes","C5.0","CART","ctree","bagging","boosting","随机森林"),errTrain_lm=rep(0,7),errTest_lm=rep(0,7),errTrain_rf=rep(0,7),errTest_rf=rep(0,7))
# 构建result.10 存放结果(10折交叉)
result.10 <-data.frame(model=c("决策树","随机森林","人工神经网络"),errTrain_lm=rep(0,3),errTest_lm=rep(0,3),errTrain_rf=rep(0,3),errTest_rf=rep(0,3))
##构建10折交叉验证
library(rpart)
control <- trainControl(method = "repeatedcv",number = 10,repeats = 3)
rpart.model.mf<- train(diabetes~.,data=indian.mf,method="rpart",trControl=control)
rpart.model.lm<- train(diabetes~.,data=indian.lm,method="rpart",trControl=control)
# 随机森林
rf.model.mf <- train(diabetes~.,data=indian.mf,method="rf",trControl=control) #8
rf.model.lm <- train(diabetes~.,data=indian.lm,method="rf",trControl=control) #2
# 神经网络
nnet.model.mf <- train(diabetes~.,data=indian.mf,method="nnet",trControl=control)
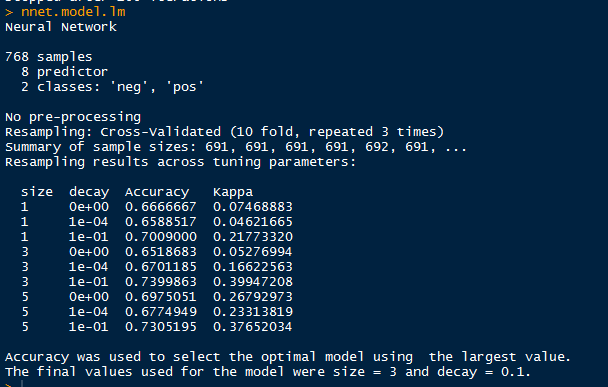
nnet.model.lm <- train(diabetes~.,data=indian.lm,method="nnet",trControl=control)
# 查看结果
rpart.model.mf
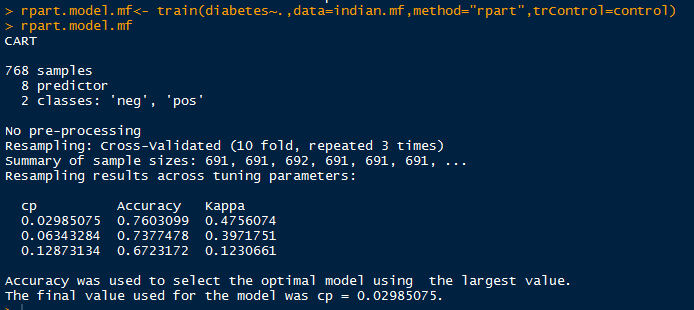
rpart.model.lm
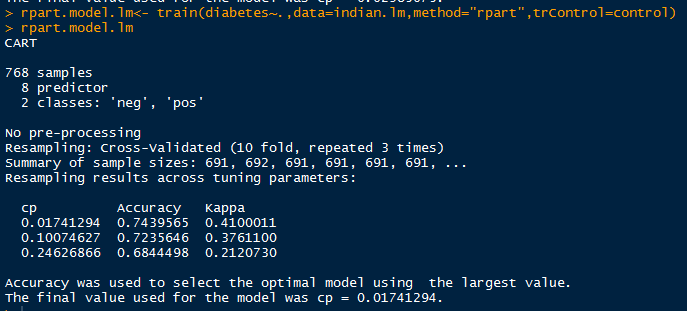
rf.model.mf

rf.model.lm
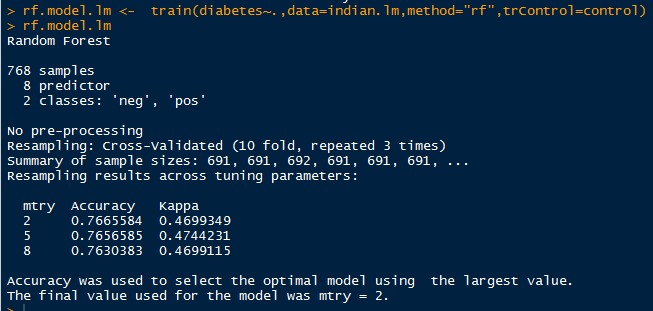
nnet.model.mf
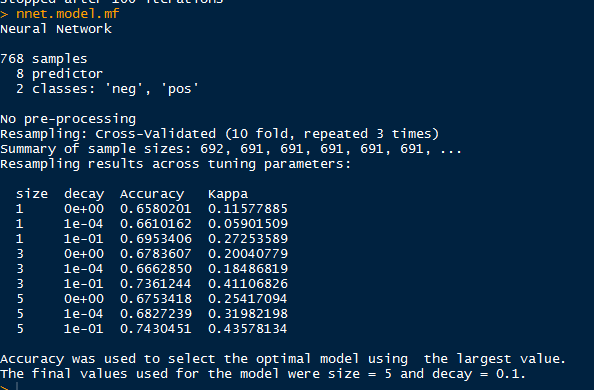
nnet.model.lm
通过循环,把两种组数据(随机森林补差,回归补差)进行普通建模,10折交叉验证获取最优参数建模,构建混淆矩阵,评估模型。
for(a in 1:2)
{
# 分别使用回归模型补差数据集\随机森林补差数据集 进行建模
ind <- createDataPartition(switch(a,indian.lm$diabetes,indian.mf$diabetes),times = 1,p=0.8,list = F)
train <- indian.lm[ind,] # 构建训练集
test <- indian.lm[-ind,] # 构建测试集
prop.table(table(indian.lm$diabetes))
prop.table(table(train$diabetes))
prop.table(table(test$diabetes))
### 建模和评估
# 使用naiveBayes函数建立朴素贝叶斯分类器
library(e1071)
str(indian.lm)
naiveBayes.model <- naiveBayes(diabetes~.,data = train) # 构建模型
# 预测结果
train_predict <- predict(naiveBayes.model,newdata=train)
test_predict <- predict(naiveBayes.model,newdata=test)
# 构建混淆矩阵
tableTrain <- table(actual=train$diabetes,predict=train_predict)
tableTest <- table(actual=test$diabetes,predict=test_predict)
# 计算误差率
errTrain <-paste0(round((sum(tableTrain)-sum(diag(tableTrain)))*100/sum(tableTrain),2),"%")
errTest <-paste0(round((sum(tableTest)-sum(diag(tableTest)))*100/sum(tableTest),2),"%")
# 决策树模型
library(C50)
C5.0.model <-C5.0(diabetes~.,data=train)
library(rpart)
rpart.model <-rpart(diabetes~.,data=train)
library(party)
ctree.model <- ctree(diabetes~.,data=train)
result[1,switch(a,2,4)] <-errTrain
result[1,switch(a,3,5)] <-errTest
for(i in 1:3){
# 预测结果
train_predict <-predict(switch(i,C5.0.model,rpart.model,ctree.model),newdata=train,type=switch(i,"class","class","response"))
test_predict <-predict(switch(i,C5.0.model,rpart.model,ctree.model),newdata=test,type=switch(i,"class","class","response"))
# 构建混淆矩阵
tableTrain <- table(actual=train$diabetes,predict=train_predict)
tableTest <- table(actual=test$diabetes,predict=test_predict)
# 计算误差率
result[i+1,switch(a,2,4)] <-paste0(round((sum(tableTrain)-sum(diag(tableTrain)))*100/sum(tableTrain),2),"%")
result[i+1,switch(a,3,5)] <-paste0(round((sum(tableTest)-sum(diag(tableTest)))*100/sum(tableTest),2),"%")
}
result
# 使用随机森林建模
library(adabag)
bagging.model <- bagging(diabetes~.,data=train)
boosting.model <-boosting(diabetes~.,data=train)
library(randomForest)
randomForest.model <- randomForest(diabetes~.,data=train)
for(i in 1:3){
# 预测结果
train_predict <-predict(switch(i,bagging.model,boosting.model,randomForest.model),newdata = train)
test_predict <- predict(switch(i,bagging.model,boosting.model,randomForest.model),newdata = test)
# 构建混淆矩阵
tableTrain <- table(actual=train$diabetes,predict=switch(i,train_predict$class,train_predict$class,train_predict))
tableTest <- table(actual=test$diabetes,predict=switch(i,test_predict$class,test_predict$class,test_predict))
# 计算误差率
result[i+4,switch(a,2,4)] <-paste0(round((sum(tableTrain)-sum(diag(tableTrain)))*100/sum(tableTrain),2),"%")
result[i+4,switch(a,3,5)] <-paste0(round((sum(tableTest)-sum(diag(tableTest)))*100/sum(tableTest),2),"%")
}
# 通过10折交叉验证选择最优参数
library(caret)
# 查看结果
rpart.model1
rf.model
nnet.model
# 利用rpart函数构建分类树
rpart.model1 <- rpart::rpart(diabetes~.,data = train,control = c(cp=switch(a,0.01741294, 0.02985075)))
# 利用randomForest函数构建随机森林
rf.model <-randomForest::randomForest(diabetes~.,data = train,mtry=switch(a,2,8))
# 利用nnet函数建立人工神经网络
nnet.model <- nnet::nnet(diabetes~.,data = train,size=switch(a,5,3),decay=0.1)
for(i in 1:3){
# 预测结果
train_predict <- predict(switch(i,rpart.model1,rf.model,nnet.model),newdata = train,type="class")
test_predict <- predict(switch(i,rpart.model1,rf.model,nnet.model),newdata = test,type="class")
# 构建混淆矩阵
tableTrain <- table(actual=train$diabetes,predict=train_predict)
tableTest <- table(actual=test$diabetes,predict=test_predict)
# 计算误差率
result.10[i,switch(a,2,4)] <-paste0(round((sum(tableTrain)-sum(diag(tableTrain)))*100/sum(tableTrain),2),"%")
result.10[i,switch(a,3,5)] <-paste0(round((sum(tableTest)-sum(diag(tableTest)))*100/sum(tableTest),2),"%")
}
}
# 查看模型评估
result
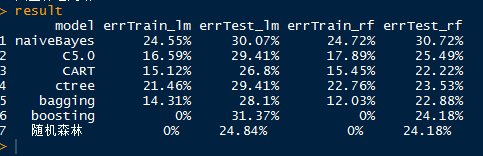
# 10折交叉验证结果
result.10
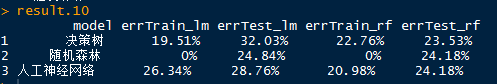
总结:通过对两组数据建模,构建混淆矩阵的结果主要可以得出如下结论:
1. 整体而言,随机森林的模型要优于其它模型
2. 随机森林中使用randomForest和boosting函数建模的所有训练集误差率为0%,测试集数据的误差率达到24~30%
3. 整体而言,当前的所有模型还没有达到最优,需要进一步探索。















