PART 1
(一) 作业要求
数据探索:利用课上讲到的列表显示缺失值和图形探究缺失数据两种方式对缺失值模式进行探究。
(二) 做题思路
利用md.pattern和aggr函数分别对缺失值进行探究。
(三) 代码及结果
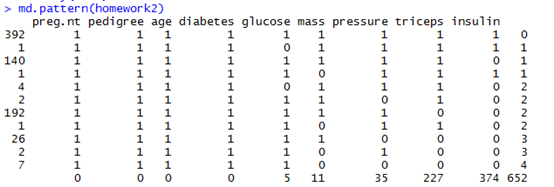
(1)用md.pattern函数以列表显示缺失值
# 列表显示缺失值
library(mice)
md.pattern(homework2)
运行结果:
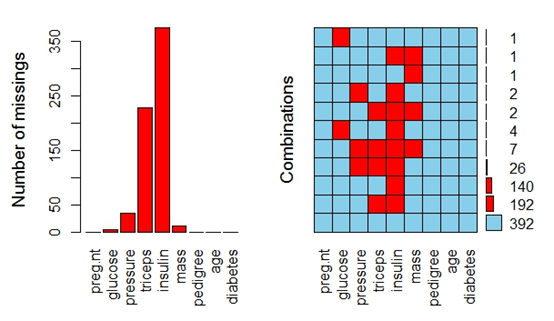
(2)用aggr函数以图形探究缺失值
# 图形探究缺失数据
library(grid)
library(VIM)
aggr(homework2,prop=FALSE,numbers=TRUE)
运行结果:
(四)结果说明
通过列表和图形的探究,在“glucose”、”pressure”、“triceps”、”insulin”、“mass”五个维度上有缺失值,其中”insulin”的缺失值最多,共有374个缺失值。
PART 2
(一)作业要求
数据完善:要求不能直接删除缺失数据,至少需要利用两种方式对缺失值进行插补。
(二)做题思路
利用随机森林和建立回归模型分别对缺失值进行插补。在建立回归模型过程中,探究最合理的模型对缺失值进行插补。
(三) 代码及结果
(1) 随机森林插补
# 随机森林插补
library(randomForest)
library(missForest)
Forest_chabu<-missForest(homework2)
Forest.full<-Forest_chabu$ximp
head(Forest.full)
运行结果:
(2) 回归模型插补
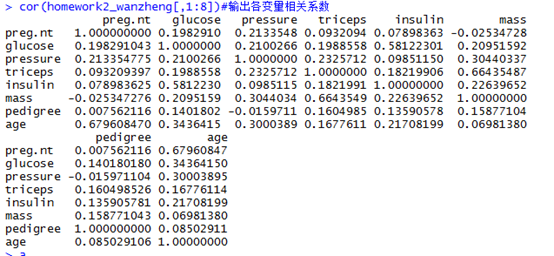
# 检验各变量相关性
ind1<-which(is.na(homework2[,2])==TRUE)
ind2<-which(is.na(homework2[,3])==TRUE)
ind3<-which(is.na(homework2[,4])==TRUE)
ind4<-which(is.na(homework2[,5])==TRUE)
ind5<-which(is.na(homework2[,6])==TRUE)
homework2_wanzheng<-homework2[-c(ind1,ind2,ind3,ind4,ind5),]
cor(homework2_wanzheng[,1:8])#输出各变量相关系数
运行结果:
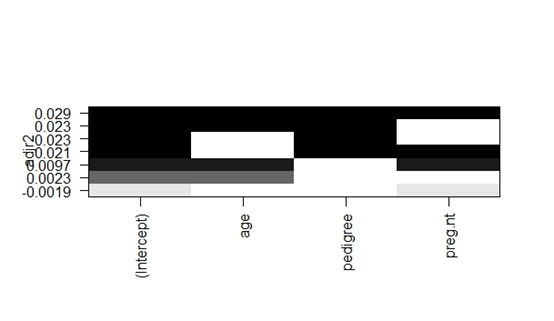
#用regsubsets函数选择合理的插补回归模型
library(leaps)
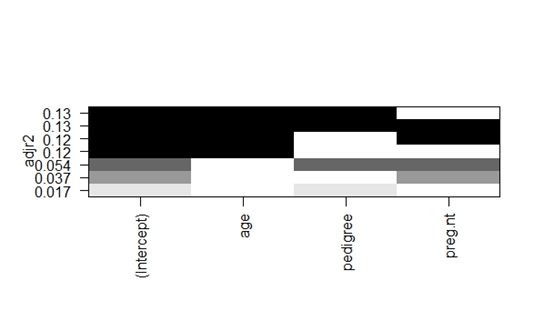
leaps1<-regsubsets(glucose~age+pedigree+preg.nt,data=homework2_wanzheng,nbest=3)
plot(leaps1,scale="adjr2")
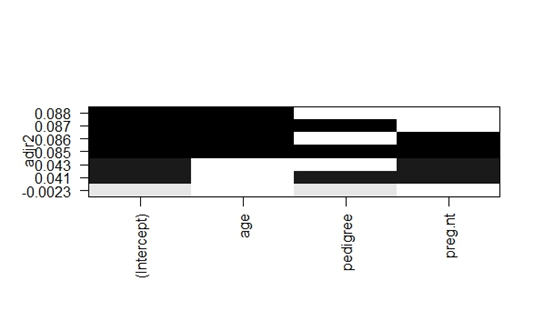
leaps2<-regsubsets(pressure~age+pedigree+preg.nt,data=homework2_wanzheng,nbest=3)
plot(leaps2,scale="adjr2")
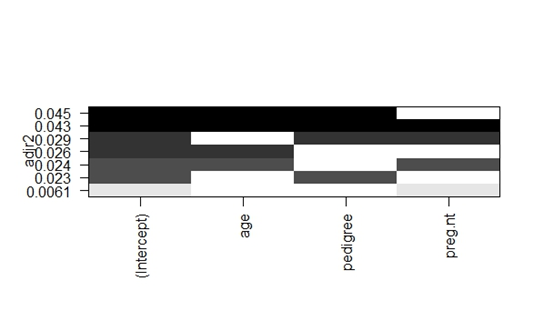
leaps3<-regsubsets(triceps~age+pedigree+preg.nt,data=homework2_wanzheng,nbest=3)
plot(leaps3,scale="adjr2")
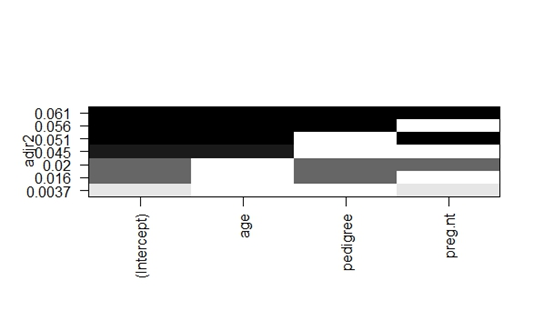
leaps4<-regsubsets(insulin~age+pedigree+preg.nt,data=homework2_wanzheng,nbest=3)
plot(leaps4,scale="adjr2")
leaps5<-regsubsets(mass~age+pedigree+preg.nt,data=homework2_wanzheng,nbest=3)
plot(leaps5,scale="adjr2")
运行结果:
#构建插补模型
dataTE1<-homework2[ind1,]
dataTE2<-homework2[ind2,]
dataTE3<-homework2[ind3,]
dataTE4<-homework2[ind4,]
dataTE5<-homework2[ind5,]
lm1<-lm(glucose~age+pedigree+preg.nt,data=homework2_wanzheng)
lm2<-lm(pressure~age+pedigree+preg.nt,data=homework2_wanzheng)
lm3<-lm(triceps~age+pedigree+preg.nt,data=homework2_wanzheng)
lm4<-lm(insulin~age+pedigree+preg.nt,data=homework2_wanzheng)
lm5<-lm(mass~age+pedigree+preg.nt,data=homework2_wanzheng)
homework2[ind1,2]=round(predict(lm1,dataTE1))#利用dataTE中数据按照模型lm对homewoork2中的缺失数据进行预测
homework2[ind2,3]=round(predict(lm2,dataTE2))
homework2[ind3,4]=round(predict(lm3,dataTE3))
homework2[ind4,5]=round(predict(lm4,dataTE4))
homework2[ind5,6]=round(predict(lm5,dataTE5))
head(homework2)
运行结果:
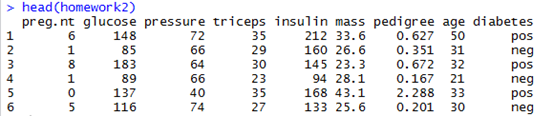
(四)结果说明
由于各变量相关度较低且用regsubsets函数探究后各类型模型R2数值均较低,最终决定确定“因变量~ age+pedigree+preg.nt”为插补模型。
PART 3
(一)作业要求
数据分区:需要按照变量diabetes来进行等比例抽样,其中80%作为训练集train数据,20%作为测试集test数据。
(二)做题思路
利用caret包中的createDataPartition函数对数据按比例抽样。
(三) 代码及结果
library(lattice)
library(caret)
ind<-createDataPartition(homework2$diabetes,times=1,p=0.8,list=F)
traindata<-homework2[ind,] #构建训练集
testdata <-homework2[-ind,] #构建测试集
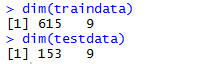
dim(traindata)
dim(testdata)
运行结果:
PART 4
(一) 作业要求
建立模型及评估:利用分类算法对train数据集建立分类预测模型,并对test数据集进行预测,利用混淆矩阵查看模型评估效果。
(二)做题思路
利用朴素贝叶斯、决策树、集成学习及随机森林、人工神经网络算法建模并评估模型效果。
(三) 代码及结果
(1) 朴素贝叶斯算法
library(e1071)
navieBayes.model<-naiveBayes(diabetes~.,data=traindata)
traindata_predict1<-predict(navieBayes.model,newdata= traindata)
testdata_predict1<-predict(navieBayes.model,newdata= testdata)
tableTR1<-table(actual=traindata$diabetes,predict=traindata_predict1)
tableTE1<-table(actual=testdata$diabetes,predict=testdata_predict1)
errTR1<-paste0(round((sum(tableTR1)-sum(diag(tableTR1)))*100/sum(tableTR1),2),"%")
errTE1<-paste0(round((sum(tableTE1)-sum(diag(tableTE1)))*100/sum(tableTE1),2),"%")
errTR1
errTE1
运行结果:

(2) 决策树算法
# 使用C50函数实现C5.0算法
library(C50)
C5.0_model<-C5.0(diabetes~.,data=traindata)
# 使用rpart函数实现CART算法
library(rpart)
rpart.model<-rpart(diabetes~.,data=traindata)
# 使用ctree函数实现条件推理决策树算法
library(mvtnorm)
library(modeltools)
library(party)
ctree.model<-ctree(diabetes~.,data=traindata)
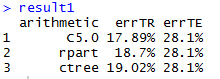
result1<-data.frame(arithmetic=c("C5.0","rpart","ctree"),errTR=rep(0,3),errTE=rep(0,3))
for(i in1:3){
traindata_predict2<-predict(switch(i,C5.0_model,rpart.model,ctree.model),newdata=traindata,type=switch(i,"class","class","response"))
testdata_predict2<-predict(switch(i,C5.0_model,rpart.model,ctree.model),newdata=testdata,type=switch(i,"class","class","response"))
tableTR2<-table(actual=traindata$diabetes,predict=traindata_predict2)
tableTE2<-table(actual=testdata$diabetes,predict=testdata_predict2)
result1[i,2]<-paste0(round((sum(tableTR2)-sum(diag(tableTR2)))*100/sum(tableTR2),2),"%")
result1[i,3]<-paste0(round((sum(tableTE2)-sum(diag(tableTE2)))*100/sum(tableTE2),2),"%")
}
result1
运行结果:
(3) 集成学习及随机森林
# 使用adabag包中的bagging函数实现bagging算法
library(mlbench)
library(adabag)
bagging.model<-bagging(diabetes~.,data=traindata)
# 使用adabag包中的boosting函数实现boosting算法
boosting.model<-boosting(diabetes~.,data=traindata)
# 使用randomForest包中的randomForest函数实现随机森林算法
library(randomForest)
randomForest.model<-randomForest(diabetes~.,data=traindata)
result2<-data.frame(arithmetic=c("bagging","boosting","randomForest"),
errTR=rep(0,3),errTE=rep(0,3))
for(i in1:3){
traindata_predict3<-predict(switch(i,bagging.model,boosting.model,randomForest.model), newdata=traindata)
testdata_predict3<-predict(switch(i,bagging.model,boosting.model,randomForest.model),newdata=testdata)
tableTR3<-table(actual=traindata$diabetes, predict=switch(i,traindata_predict3$class,traindata_predict3$class,traindata_predict3))
tableTE3<-table(actual=testdata$diabetes,predict=switch(i,testdata_predict3$class,testdata_predict3$class,testdata_predict3))
result2[i,2]<-paste0(round((sum(tableTR3)-sum(diag(tableTR3)))*100/sum(tableTR3),2),"%")
result2[i,3]<-paste0(round((sum(tableTE3)-sum(diag(tableTE3)))*100/sum(tableTE3),2),"%")
}
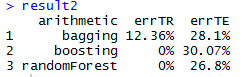
result2
运行结果:
(4) 人工神经网络与支持向量机
# 使用nnet包中的nnet函数建立人工神经网络模型
library(nnet)
nnet.model<- nnet(diabetes~.,data=traindata,size=3,rang=0.1,decay=5e-4,maxit=200)
# 使用kernlab包中的ksvm函数建立支持向量机模型
library(kernlab)
# 使用rbfdot选项制定径向基核函数
svm.model<- ksvm(diabetes~.,data=traindata,kernel="rbfdot")
# 利用模型对结果进行预测
# 构建混淆矩阵
nnet.t0 <-table(traindata$diabetes,predict(nnet.model,traindata,type="class"))
nnet.t <-table(testdata$diabetes,predict(nnet.model,testdata,type="class"))
svm.t0 <-table(traindata$diabetes,predict(svm.model,traindata,type="response"))
svm.t <-table(testdata$diabetes,predict(svm.model,testdata,type="response"))
# 查看各自错误率
nnet.err0<- paste0(round((sum(nnet.t0)-sum(diag(nnet.t0)))*100/sum(nnet.t0),2),"%")
nnet.err<- paste0(round((sum(nnet.t)-sum(diag(nnet.t)))*100/sum(nnet.t),2),"%")
svm.err0<- paste0(round((sum(svm.t0)-sum(diag(svm.t0)))*100/sum(svm.t0), 2),"%")
svm.err <-paste0(round((sum(svm.t)-sum(diag(svm.t)))*100/sum(svm.t),2),"%")
nnet.err0;nnet.err;svm.err0;svm.err
运行结果:

(四)结果说明
经过对比后,模型相对合适的为通过ksvm函数建立的支持向量机模型,训练集与测试集的误差率分别为19.19%与24.84%。值得注意的是,用boosting与随机森林建立的模型在训练集误差率为0%。但总体来说,几个模型的总体误差率都较大。