本文作者 未禾,首发于作者知乎,https://zhuanlan.zhihu.com/p/27570774,已获作者授权原创形式发布,欢迎点击【阅读原文】关注支持!
第三章 分布数据集的可视化
在处理一组数据时,通常首先要做的是了解变量是如何分布的。这一章将简要介绍seborn中用于检查单变量和双变量分布的一些工具。你可能还想看看分类变量的章节,来看看函数的例子,这些函数让我们很容易比较变量的分布。
%matplotlib inline
import numpy as np
import pandas as pd
from scipy import stats, integrate
import matplotlib.pyplot as plt
import seaborn as sns
sns.set(color_codes=True)
np.random.seed(sum(map(ord, "distributions")))
单变量分布
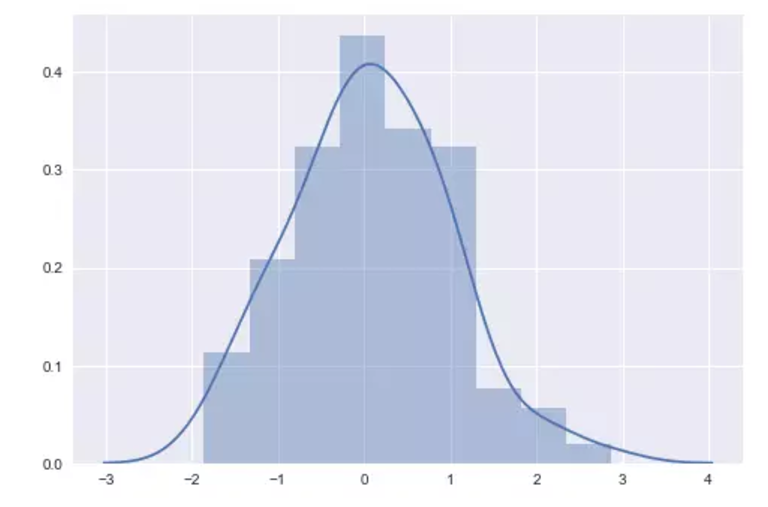
最方便的方式是快速查看单变量分布无疑是使用distplot()函数。默认情况下,这将绘制一个直方图,并拟合出核密度估计(KDE)。
x = np.random.normal(size=100)
sns.distplot(x);
直方图
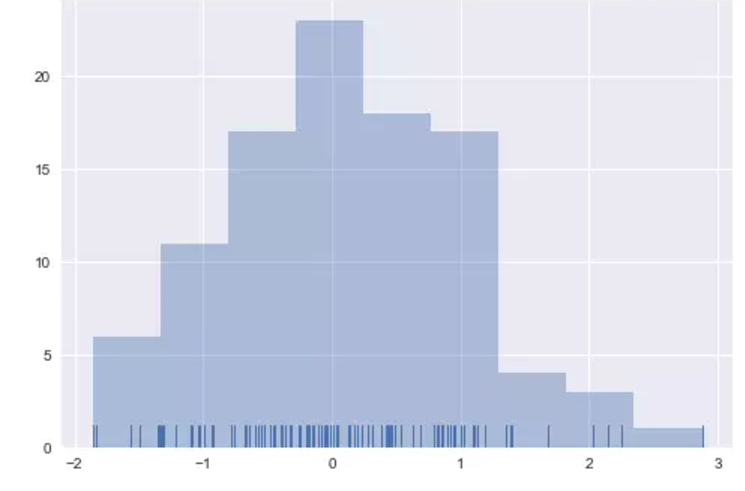
直方图应当是非常熟悉的函数了,在matplotlib中就存在hist函数。直方图通过在数据的范围内切成数据片段,然后绘制每个数据片段中的观察次数,来表示整体数据的分布。
为了说明这一点,我们删除密度曲线并添加了地毯图,每个观察点绘制一个小的垂直刻度。您可以使用rugplot()函数来制作地毯图,但它也可以在distplot()中使用:
sns.distplot(x, kde=False, rug=True);
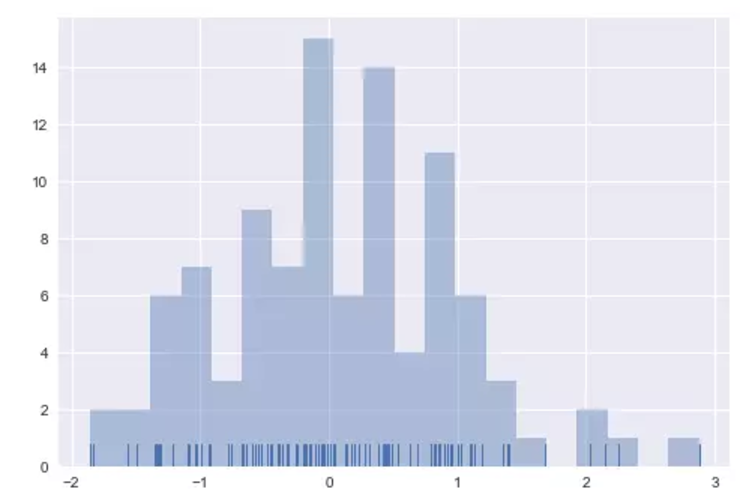
绘制直方图时,主要的选择是使用切分数据片段的数量或在何位置切分数据片段。 distplot()使用一个简单的规则来很好地猜测并给予默认的切分数量,但尝试更多或更少的数据片段可能会显示出数据中的其他特征:
sns.distplot(x, bins=20, kde=False, rug=True);
核密度估计(KDE)
或许你对核密度估计(KDE,Kernel density estimaton)可能不像直方图那么熟悉,但它是绘制分布形状的有力工具。如同直方图一样,KDE图会对一个轴上的另一轴的高度的观测密度进行描述:
sns.distplot(x, hist=False, rug=True);
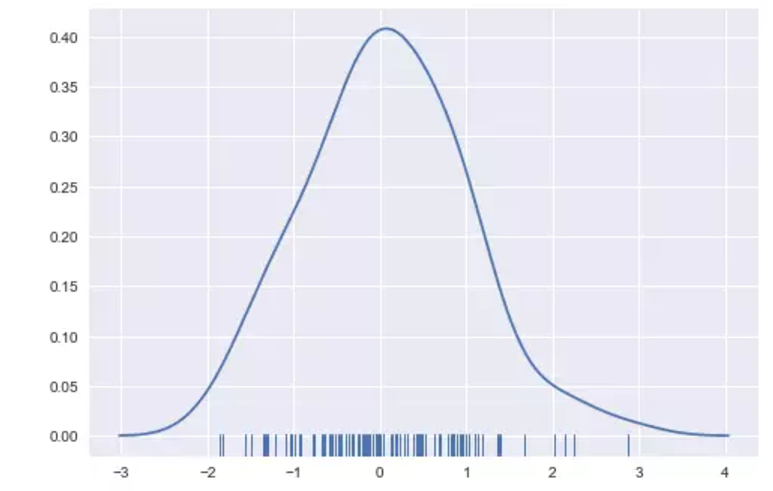
绘制KDE比绘制直方图更有计算性。所发生的是,每一个观察都被一个以这个值为中心的正态( 高斯)曲线所取代。
x = np.random.normal(0, 1, size=30)
bandwidth = 1.06 * x.std() * x.size ** (-1 / 5.)
support = np.linspace(-4, 4, 200)
kernels = []
for x_i in x:
kernel = stats.norm(x_i, bandwidth).pdf(support)
kernels.append(kernel)
plt.plot(support, kernel, color="r")
sns.rugplot(x, color=".2", linewidth=3);
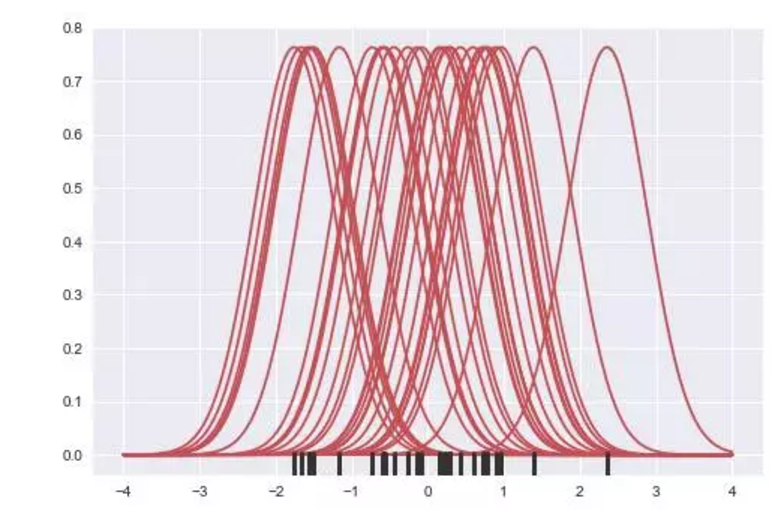
接下来,这些曲线可以用来计算支持网格中每个点的密度值。得到的曲线再用归一化使得它下面的面积等于1:
density = np.sum(kernels, axis=0)
density /= integrate.trapz(density, support)
plt.plot(support, density);
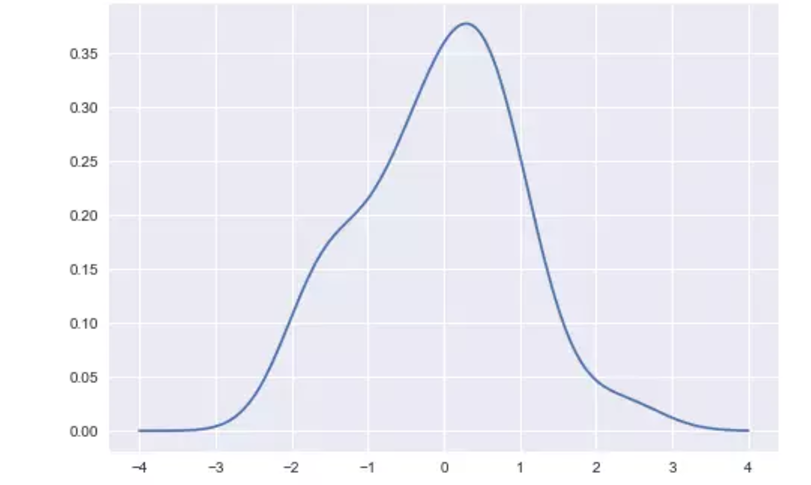
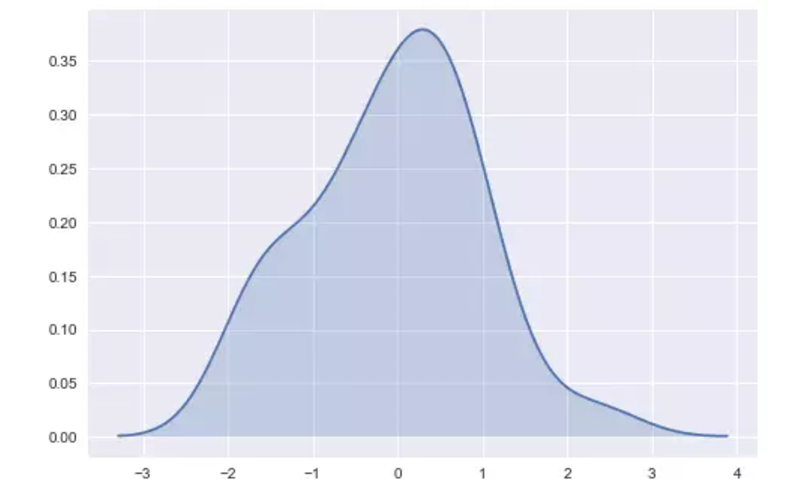
我们可以看到,如果我们在seaborn中使用kdeplot()函数,我们得到相同的曲线。 这个函数由distplot()使用,但是当您只想要密度估计时,它提供了一个更直接的界面,更容易访问其他选项:
sns.kdeplot(x, shade=True);
KDE的带宽bandwidth(bw)参数控制估计对数据的拟合程度,与直方图中的bin(数据切分数量参数)大小非常相似。 它对应于我们上面绘制的内核的宽度。 默认中会尝试使用通用引用规则猜测一个适合的值,但尝试更大或更小的值可能会有所帮助:
sns.kdeplot(x)
sns.kdeplot(x, bw=.2, label="bw: 0.2")
sns.kdeplot(x, bw=2, label="bw: 2")
plt.legend();
如上所述,高斯KDE过程的性质意味着估计延续了数据集中最大和最小的值。 可以通过cut参数来控制绘制曲线的极值值的距离; 然而,这只影响曲线的绘制方式,而不是曲线如何拟合:
sns.kdeplot(x, shade=True, cut=0)
sns.rugplot(x);
拟合参数分布
还可以使用distplot()将参数分布拟合到数据集,并可视化地评估其与观察数据的对应关系:
x = np.random.gamma(6, size=200)
sns.distplot(x, kde=False, fit=stats.gamma);
绘制双变量分布
在绘制两个变量的双变量分布也是有用的。在seaborn中这样做的最简单的方法就是在jointplot()函数中创建一个多面板数字,显示两个变量之间的双变量(或联合)关系以及每个变量的单变量(或边际)分布和轴。
mean, cov = [0, 1], [(1, .5), (.5, 1)]
data = np.random.multivariate_normal(mean, cov, 200)
df = pd.DataFrame(data, columns=["x", "y"])
散点图
双变量分布的最熟悉的可视化方式无疑是散点图,其中每个观察结果以x和y值表示。这是两个方面的地毯图。可以使用matplotlib中的plt.scatter函数绘制散点图,它也是jointplot()函数显示的默认方式。
sns.jointplot(x="x", y="y", data=df);
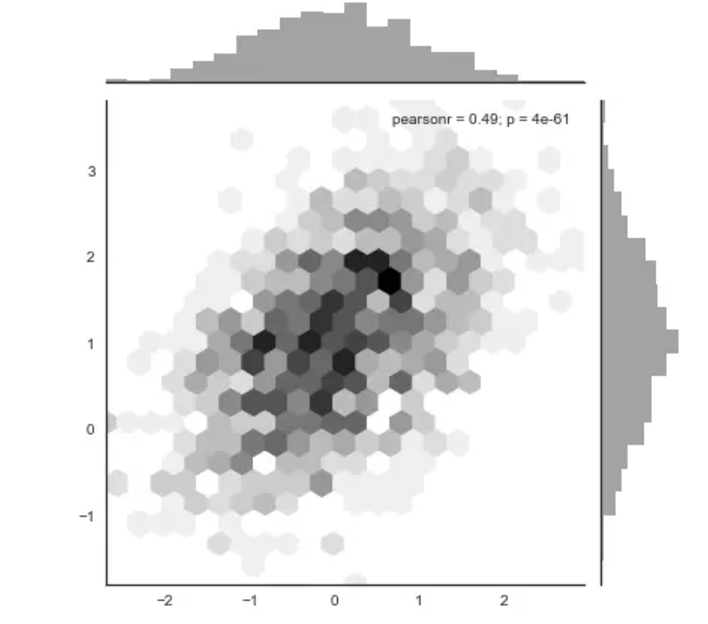
HexBin图
直方图的双变量类似物被称为“hexbin”图,因为它显示了落在六边形仓内的观测数。该图适用于较大的数据集。通过matplotlib plt.hexbin函数和jointplot()中的样式可以实现。 它最好使用白色背景:
x, y = np.random.multivariate_normal(mean, cov, 1000).T
with sns.axes_style("white"):
sns.jointplot(x=x, y=y, kind="hex", color="k");
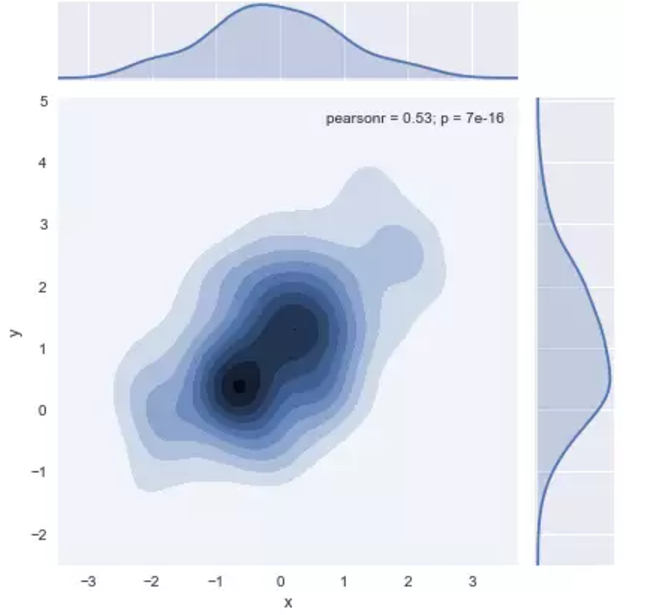
核密度估计
使用上述内核密度估计程序可视化双变量分布也是可行的。在seaborn中,这种图用等高线图显示,可以在jointplot()中作为样式传入参数使用:
sns.jointplot(x="x", y="y", data=df, kind="kde");
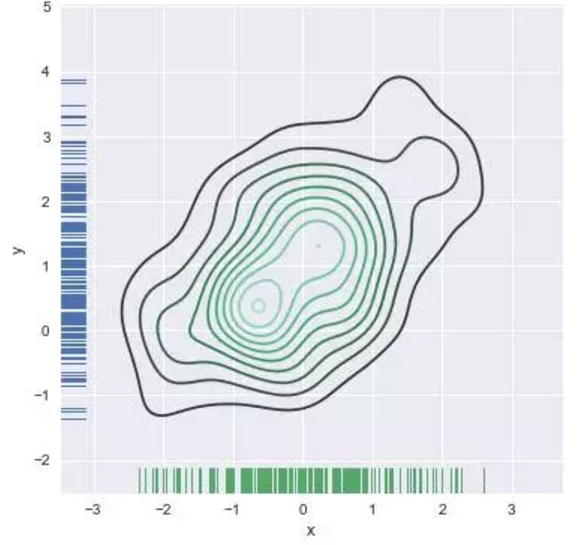
还可以使用kdeplot()函数绘制二维核密度图。这样可以将这种绘图绘制到一个特定的(可能已经存在的)matplotlib轴上,而jointplot()函数只能管理自己:
f, ax = plt.subplots(figsize=(6, 6))
sns.kdeplot(df.x, df.y, ax=ax)
sns.rugplot(df.x, color="g", ax=ax)
sns.rugplot(df.y, vertical=True, ax=ax);
如果是希望更连续地显示双变量密度,您可以简单地增加n_levels参数增加轮廓级数:
f, ax = plt.subplots(figsize=(6, 6))
cmap = sns.cubehelix_palette(as_cmap=True, dark=0, light=1, reverse=True)
sns.kdeplot(df.x, df.y, cmap=cmap, n_levels=60, shade=True);

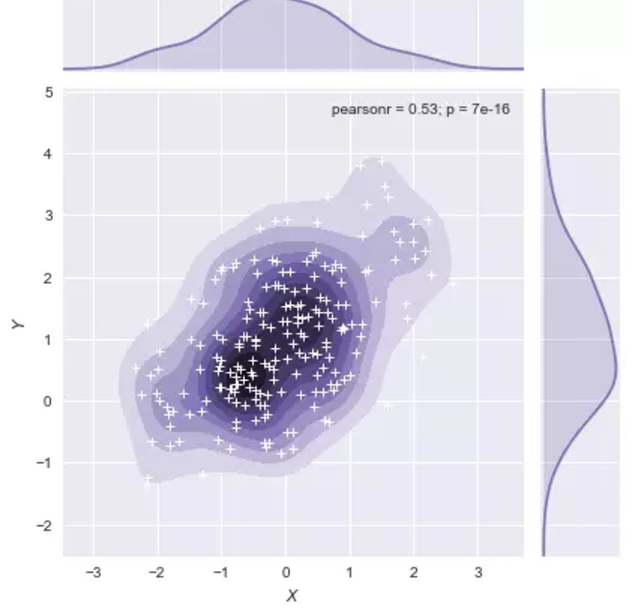
jointplot()函数使用JointGrid来管理。为了获得更多的灵活性,您可能需要直接使用JointGrid绘制图形。jointplot()在绘制后返回JointGrid对象,您可以使用它来添加更多图层或调整可视化的其他方面:
g = sns.jointplot(x="x", y="y", data=df, kind="kde", color="m")
g.plot_joint(plt.scatter, c="w", s=30, linewidth=1, marker="+")
g.ax_joint.collections[0].set_alpha(0)
g.set_axis_labels("$X$", "$Y$");
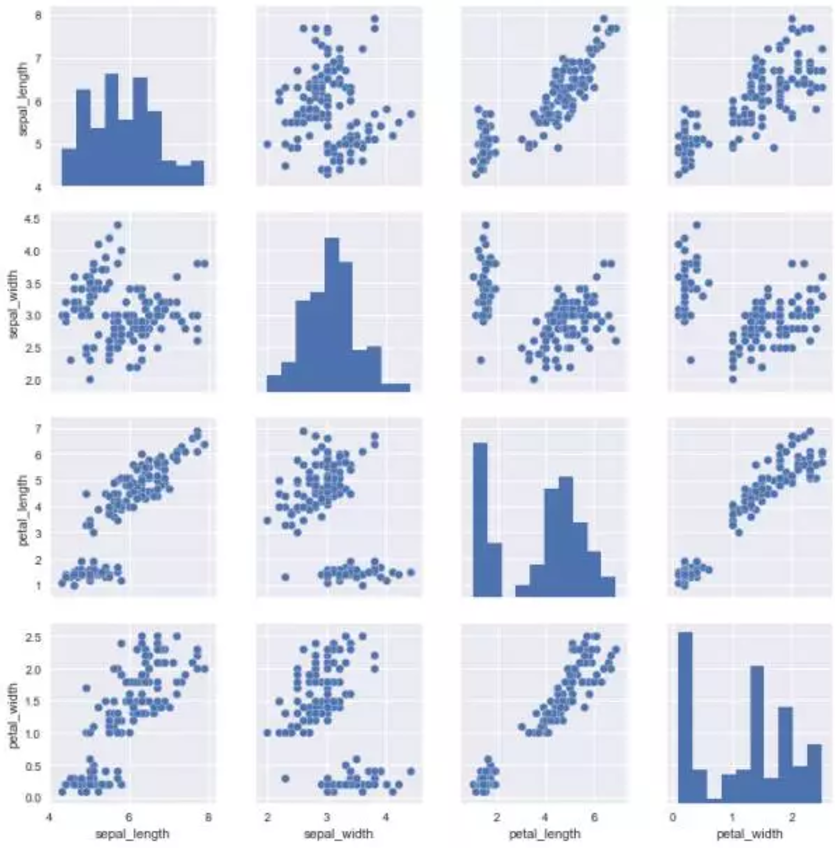
呈现数据集中成对的关系
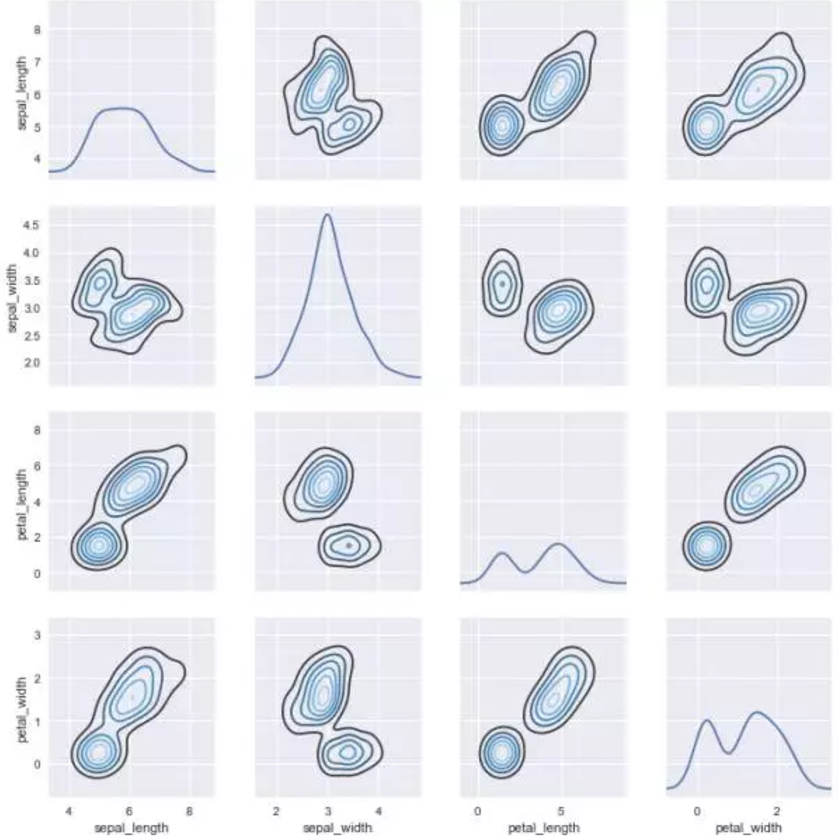
要在数据集中绘制多个成对双变量分布,可以使用pairplot()函数。这将创建一个轴的矩阵,并显示DataFrame中每对列的关系。默认情况下,它也绘制每个变量在对角轴上的单变量:
iris = sns.load_dataset("iris")
sns.pairplot(iris);
对于jointplot()和JointGrid之间的关系,pairplot()函数是建立在一个PairGrid对象上的,可以直接使用它来获得更大的灵活性:
g = sns.PairGrid(iris)
g.map_diag(sns.kdeplot)
g.map_offdiag(sns.kdeplot, cmap="Blues_d", n_levels=6);