本文的主要内容就是针对经典的Breast Cancer Wisconsin (Diagnostic)数据,分别通过R和Python两种语言去实现KNN分类算法。
本文的R代码源于Brett Lantz的《Machine Learning with R》书籍第三章。当然这本书大家也不少人看过,并且也看过很多的有关这个案例的分析,今天我就给大家用两种语言去对比实现此案例的实战。
文章共分为四部分:
(1)加载程序包
(1)读取数据
(2)探索和准备数据
(3)训练模型
(4)模型评估
## 加载程序包
#### R code
library(caret) # preProcess
library(class) # knn
#### Py code
import pandas as pd #read_csv
from sklearn.preprocessing import MinMaxScaler
from sklearn.neighbors import KNeighborsClassifier
from sklearn.metrics import confusion_matrix
在R中一共加载了两个程序包,代码后面注释代表后续用到的此包中的函数。Py中就不用解释了,代码就已经很清楚了。
## 读取数据
#### R code
wisconsin <- read.csv("../input/wisconsin.csv")
#### Py code
wisconsin = pd.read_csv("../input/wisconsin.csv")
## 探索和准备数据
## 整体探索数据信息
#### R code
str(wisconsin)
head(wisconsin)
summary(wisconsin)
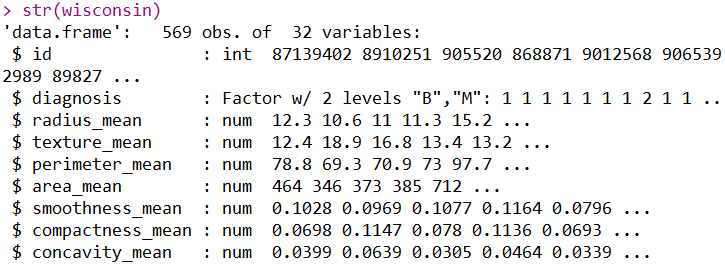
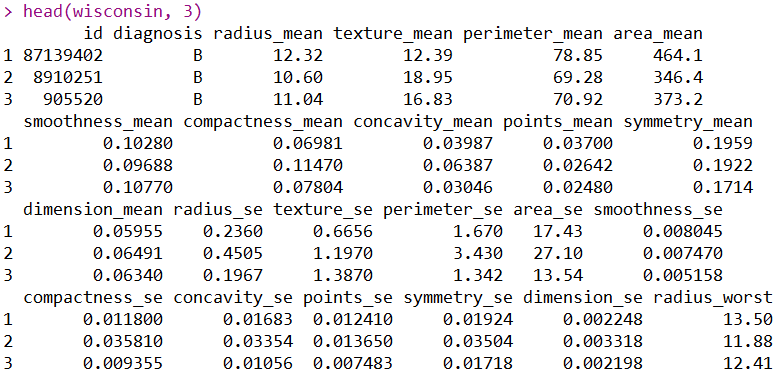
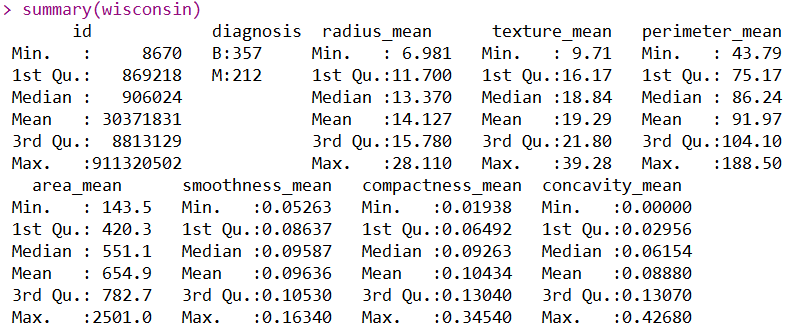
以上分别是R对数据集简单结构的探索、查看数据的前3行和统计数据的主要描述性统计量。这三个操作能展现出的信息我就不在重复了,大家都这个数据集肯定也是烂熟于心了。下面使用Python语言去实现R展示出的信息:
#### Py code
wisconsin.dtypes
wisconsin.shape
wisconsin.head(3)
wisconsin.describe()
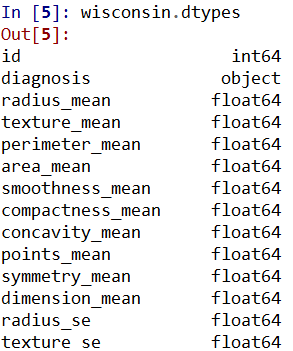
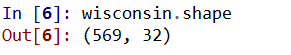

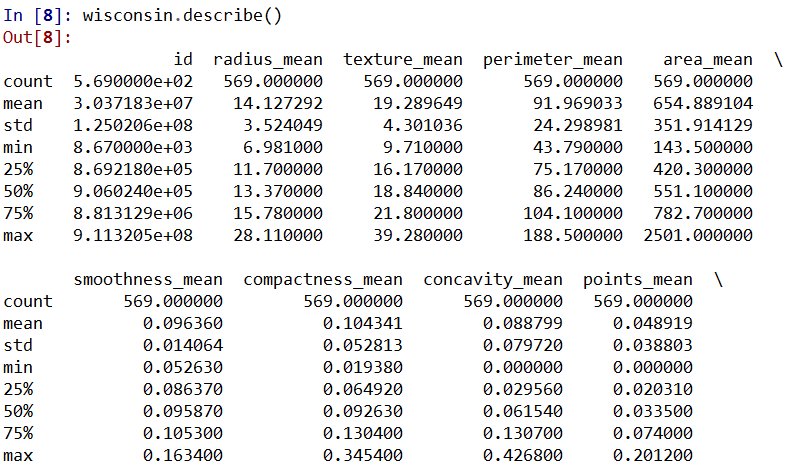
利用这四行代码,我们展示出了与R所展示出的一样的信息。
## 探索数据的目标变量
#### R code
wisconsin <- wisconsin[-1]
table(wisconsin$diagnosis)
prop.table(table(wisconsin$diagnosis))

上图三行代码的意义分别是:剔除数据集的第一列("id"),此列对于建模没有任何意义;查看目标变量("diagnosis")的特征值的个数;查看目标变量("diagnosis")的特征值的占比。
#### Py code
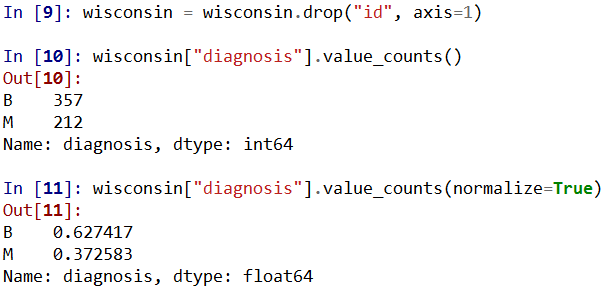
wisconsin = wisconsin.drop("id", axis=1)
wisconsin["diagnosis"].value_counts()
wisconsin["diagnosis"].value_counts(normalize=True)
## 对自变量进行归一化
#### R code
normalize <- function(x) {
return( (x-min(x)) / (max(x)-min(x)))
}
wisconsin[2:31] <- as.data.frame(lapply(wisconsin[2:31], normalize))
# standard <- preProcess(wisconsin, method = 'range')
# wisconsin <- predict(standard, wisconsin))
通过自定义归一化的函数,加上使用lapply函数,我们就高效地对数据的每一列进行了归一化,从而使我们的数值都压缩到了0-1范围内。下面注释的代码是用caret包里面的函数进行标准化的,两种方法得到的数据一样,但是强烈推荐后者,因为其可以自动避免非数值型数据,从而对数值型数据进行归一化。下面我们使用py来对比一下:
#### Py code
wisconsin_x = wisconsin.drop(['diagnosis'], axis=1)
wisconsin_y = wisconsin['diagnosis']
wisconsin_sta = (wisconsin_x - wisconsin_x.min()) / (wisconsin_x.max() - wisconsin_x.min())
wisconsin_sta.head(3)
# wisconsin_sta = pd.DataFrame(MinMaxScaler().fit_transform(wisconsin_x), columns=wisconsin_x.columns)
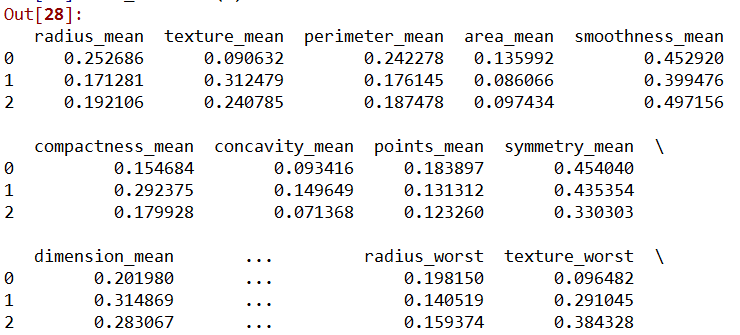
通过以上的py代码,我们同样对数据进行了归一化,后面注释的代码同样也可以达到归一化的目的。
## 建立训练集和测试集
#### R code
traindata <- wisconsin[1:469, -1]
testdata <- wisconsin[470:569, -1]
train_label <- wisconsin[1:469, 1]
test_label <- wisconsin[470:569, 1]
#### Py code
traindata = wisconsin_x.loc[0:468]
testdata = wisconsin_x.loc[469:568]
train_label = wisconsin_y.loc[0:468]
test_label = wisconsin_y.loc[469:568]
上面分别使用R和Py创建了训练集、测试集和训练集标签、测试集标签。下面开始使用knn进行分类:
## 训练模型
#### R code
pred <- knn(traindata, testdata, cl = train_label, k = 21)
#### Py code
clf = KNeighborsClassifier(n_neighbors=21)
clf = clf.fit(X=traindata, y=train_label)
pred = clf.predict(testdata)
上面分别使用R和Py使用训练集建立了knn模型,下面使用混淆矩阵做模型评估:
## 模型评估
#### R code
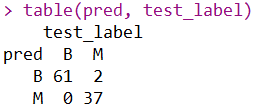
table(pred, test_label)
#### Py code
confusion_matrix(test_label, pred)

以上是混淆矩阵,大家也看到了混淆矩阵中的“2”处的位置不同,可能会有疑问。这是因为在建立混淆矩阵的时候,py的参数是先写真实值,后写预测值,所以说其标签也会有相应的倒置。这个混淆矩阵的结果意味着,在100个测试集中,有两个属于“M”(恶性)的细胞被错误预测为了“B”(良性)。
注:本案例不提供数据集,如果要学习完整案例,点击文章底部阅读原文或者扫描课程二维码,购买包含数据集+代码+PPT的《kaggle十大案例精讲课程》,购买学员会赠送文章的数据集。
《kaggle十大案例精讲课程》提供代码+数据集+详细代码注释+老师讲解PPT!综合性的提高你的数据能力,数据处理+数据可视化+建模一气呵成!
