往期回顾
前言
在上一期中,关于线性回归模型的创建,我们对比了Python和R语言的具体代码实现,受到了很多网友的关注。也有一些朋友问到,关于线性回归模型的那些前提假设为什么没有作分享,这期和下期我们就来回答一下这个问题。由于线性回归模型的偏回归系数通过最小二乘法(OLS)实现的,关于最小二乘法的使用是有一些假设前提的,具体是:
自变量与因变量之间存在线性关系;
自变量之间不存在多重共线性;
回归模型的残差服从正态分布;
回归模型的残差满足方差齐性(即方差为某个固定值);
回归模型的残差之间互相独立;
当然,除了上面提到的5点之外,我们还需要注意的是,线性回归模型对异常值是非常敏感的,模型预测的结果非常容易受到异常值的影响,所以,我们还需要对原始数据进行异常点识别和处理。在本期中,我们先针对上面提到的前三个假设做判别,此外再分享一下关于异常点的相关内容。本次分享的数据集来自于UCI【高炉煤气联合循环发电(CCPP)数据集(数据来源:http://archive.ics.uci.edu/ml/datasets/combined+cycle+power+plant)】。
线性性检验
线性回归模型,顾名思义,首先要保证自变量与因变量之间存在线性关系。关于线性关系的判断,我们可以通过图形或Pearson相关系数来识别,具体Python代码如下:
# 导入第三方包
import pandas as pd
import numpy as np
from patsy import dmatrices
from statsmodels.stats.outliers_influence import variance_inflation_factor
import statsmodels.api as sm
import scipy.stats as stats
from sklearn.metrics import mean_squared_error
import seaborn as sns
import matplotlib.pyplot as plt
import matplotlib.mlab as mlab
# 数据读取
ccpp = pd.read_excel('CCPP.xlsx')
ccpp.describe()
# AT:温度
# V:压力
# AP:相对湿度
# RH:排气量
# PE:发电量
# 绘制各变量之间的散点图
sns.pairplot(ccpp)
plt.show()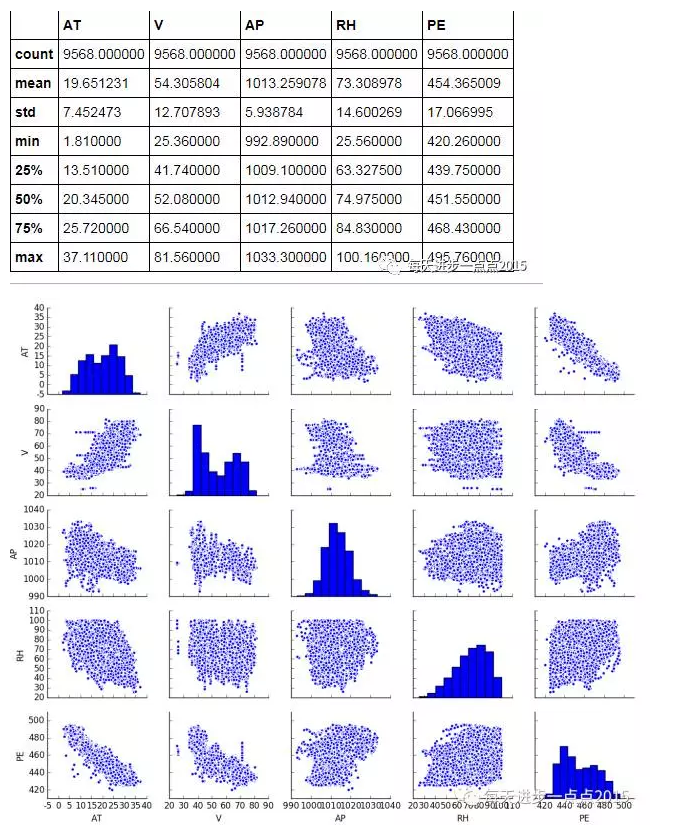
从上面的散点图来看,似乎AP(相对湿度)和RH(排气量)与PE(发电量)之间并不存在明显的线性关系,具体我们还需要看一下PE与其余变量之间的Pearson相关系数值。
# 发电量与自变量之间的相关系数
ccpp.corrwith(ccpp.PE)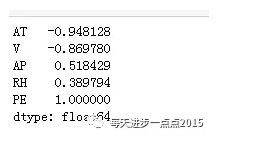
从返回的结果来看,PE(发电量)与AT(温度)和V(压力)之间的相关系数还是蛮高的,而PE与AP(相对湿度)和RH(排气量)之间的相关系数就明显小很多。一般情况下,当Pearson相关系数低于0.4,则表明变量之间存在弱相关关系;当Pearson相关系数在0.4~0.6之间,则说明变量之间存在中度相关关系;当相关系数在0.6以上时,则反映变量之间存在强相关关系。经过对比发现,PE与RH之间的为弱相关关系,故不考虑将该变量纳入模型。当然,变量之间不存在线性关系并不代表不存在任何关系,可能是二次函数关系、对数关系等,所以一般还需要进行检验和变量转换。
多重共线性检验
先来了解一下,如果模型的自变量之间存在严重的多重共线性的话,会产生什么后果呢?
导致OLS估计量可能无效;
增大OLS估计量的方差;
变量的显著性检验将失去意义;
模型缺乏稳定性;
所以,多重共线性检验就显得非常重要了,关于多重共线性的检验可以使用方差膨胀因子(VIF)来鉴定,如果VIF大于10,则说明变量存在多重共线性。一旦发现变量之间存在多重共线性的话,可以考虑删除变量和重新选择模型(岭回归法)。
# 将因变量PE,自变量AT,V,AP和截距项(值为1的1维数组)以数据框的形式组合起来
y, X = dmatrices('PE~AT+V+AP', data = ccpp, return_type='dataframe')
# 构造空的数据框
vif = pd.DataFrame()
vif["VIF Factor"] = [variance_inflation_factor(X.values, i) for i in range(X.shape[1])]
vif["features"] = X.columns
vif
结果显示,所有自变量的VIF均低于10,说明自变量之间并不存在多重共线性的隐患。
异常点检测
在异常点检测之前,我们需要对现有的数据,进行线性回归模型的构造。具体操作如下,与上一期文章中回归模型的建模一致:
# 构造PE与AT、V和AP之间的线性模型
fit = sm.formula.ols('PE~AT+V+AP',data = ccpp).fit()
fit.summary()
# 计算模型的RMSE值
pred = fit.predict()
np.sqrt(mean_squared_error(ccpp.PE, pred))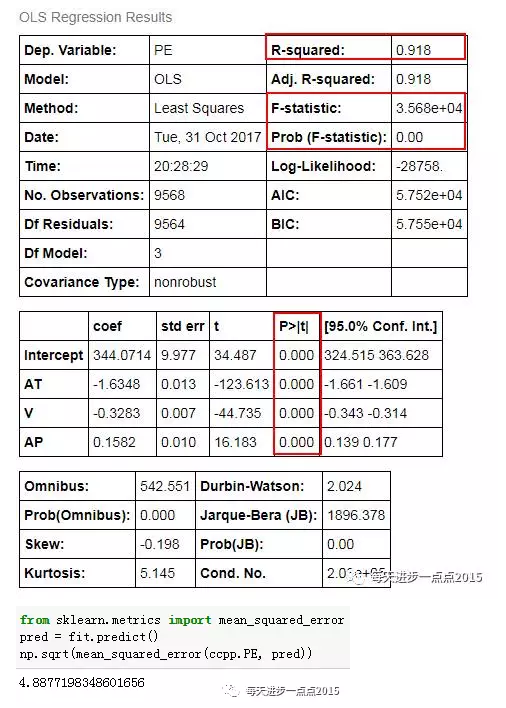
通过上面的建模结果来看,一切都显得那么的完美(模型的显著性检验通过,偏回归系数的显著性检验通过;R方值也达到了0.9以上)。尽管这样,我们还是需要基于这个模型,完成异常点的检测。
关于异常点的检测方法,一般可以通过高杠杆值点(帽子矩阵)或DFFITS值、学生化残差、cook距离和covratio值来判断。这些值的具体计算脚本如下:
# 离群点检验
outliers = fit.get_influence()
# 高杠杆值点(帽子矩阵)
leverage = outliers.hat_matrix_diag
# dffits值
dffits = outliers.dffits[0]
# 学生化残差
resid_stu = outliers.resid_studentized_external
# cook距离
cook = outliers.cooks_distance[0]
# covratio值
covratio = outliers.cov_ratio
# 将上面的几种异常值检验统计量与原始数据集合并
contat1 = pd.concat([pd.Series(leverage, name = 'leverage'),pd.Series(dffits, name = 'dffits'),
pd.Series(resid_stu,name = 'resid_stu'),pd.Series(cook, name = 'cook'),
pd.Series(covratio, name = 'covratio'),],axis = 1)
ccpp_outliers = pd.concat([ccpp,contat1], axis = 1)
ccpp_outliers.head()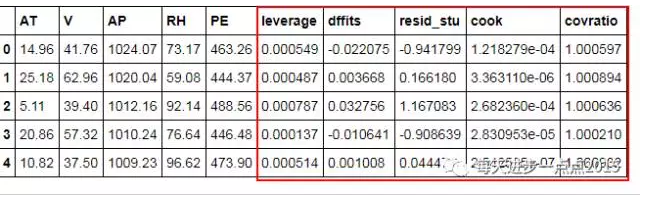
通过参考薛毅老师的《统计建模与R软件》书可知,当高杠杆值点(或帽子矩阵)大于2(p+1)/n时,则认为该样本点可能存在异常(其中p为自变量的个数,n为观测的个数);当DFFITS统计值大于2sqrt((p+1)/n)时 ,则认为该样本点可能存在异常;当学生化残差的绝对值大于2,则认为该样本点可能存在异常;对于cook距离来说,则没有明确的判断标准,一般来说,值越大则为异常点的可能性就越高;对于covratio值来说,如果一个样本的covratio值离数值1越远,则认为该样本越可能是异常值。这里我们就以学生化残差作为评判标准,因为其包含了帽子矩阵和DFFITS的信息。
# 计算异常值数量的比例
outliers_ratio = sum(np.where((np.abs(ccpp_outliers.resid_stu)>2),1,0))/ccpp_outliers.shape[0]
outliers_ratio
# 删除异常值
ccpp_outliers = ccpp_outliers.loc[np.abs(ccpp_outliers.resid_stu)<=2,]结果显示,确实存在异常值点,且异常值的数量占了3.7%。对于异常值的处理,我们可以考虑下面几种办法:
当异常比例极低时(如5%以内),可以考虑直接删除;
当异常比例比较高时,可以考虑将异常值衍生为哑变量,即异常值对应到1,非异常值则对应到0;
将单独把异常值提取出来,另行建模;
这里为了简单起见,我们将3.7%的异常值作删除处理。
# 重新建模
fit2 = sm.formula.ols('PE~AT+V+AP',data = ccpp_outliers).fit()
fit2.summary()
# 计算模型的RMSE值
pred2 = fit2.predict()
np.sqrt(mean_squared_error(ccpp_outliers.PE, pred2))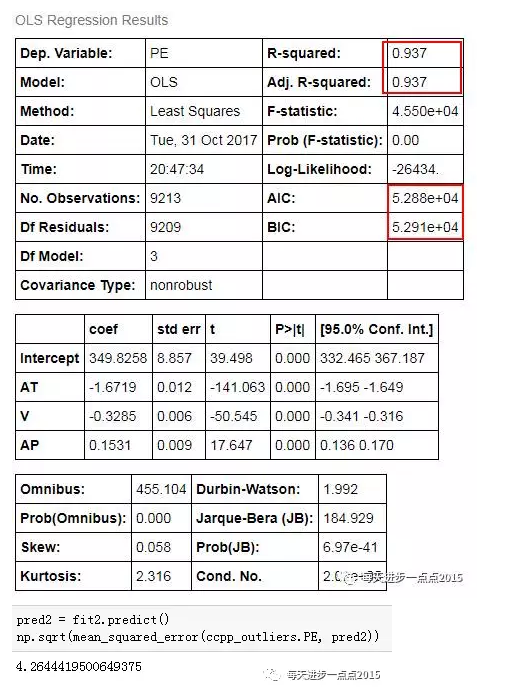
通过对比fit和fit2,将异常值删除后重新建模的话,效果会更理想一点,具体表现为:信息准则(AIC和BIC)均变小,同时RMSE(误差均方根)也由原来的4.89降低到4.26。
正态性检验
当模型的残差服从正态性假设时,才能保证模型偏回归系数对于的t值和模型的F值是有效的。故模型建好后,要对模型的残差进行正态性检验。关于正态性检验由两类方法,即定性的图形法(直方图、PP图和QQ图)和定量的非参数法(Shapiro检验和K-S检验)。
# 残差的正态性检验(直方图法)
resid = fit2.resid
# 中文和负号的正常显示
plt.rcParams['font.sans-serif'] = ['Microsoft YaHei']
plt.rcParams['axes.unicode_minus'] = False
plt.hist(resid, # 绘图数据
bins = 100, # 指定直方图条的个数
normed = True, # 设置为频率直方图
color = 'steelblue', # 指定填充色
edgecolor = 'k') # 指定直方图的边界色
# 设置坐标轴标签和标题
plt.title('残差直方图')
plt.ylabel('密度值')
# 生成正态曲线的数据
x1 = np.linspace(resid.min(), resid.max(), 1000)
normal = mlab.normpdf(x1, resid.mean(), resid.std())
# 绘制正态分布曲线
plt.plot(x1,normal,'', linewidth = 2, label = '正态分布曲线')
# 生成核密度曲线的数据
kde = mlab.GaussianKDE(resid)
x2 = np.linspace(resid.min(), resid.max(), 1000)
# 绘制核密度曲线
plt.plot(x2,kde(x2),'', linewidth = 2, label = '核密度曲线')
# 去除图形顶部边界和右边界的刻度
plt.tick_params(top='off', right='off')
# 显示图例
plt.legend(loc='best')
# 显示图形
plt.show()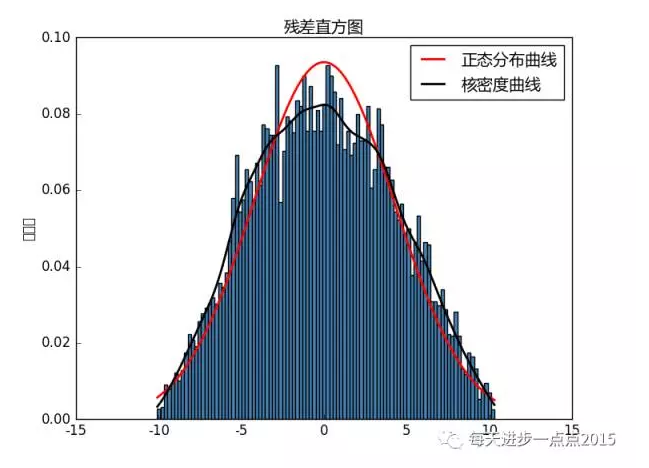
从残差的直方图来看,核密度曲线与理论的正态分布曲线的趋势还是比较吻合的,即使残差不服从正态分布,也能反映其基本近似于正态分布。
# 残差的正态性检验(PP图和QQ图法)
pp_qq_plot = sm.ProbPlot(resid)
pp_qq_plot.ppplot(line = '45')
plt.title('P-P图')
pp_qq_plot.qqplot(line = 'q')
plt.title('Q-Q图')# 显示图形plt.show()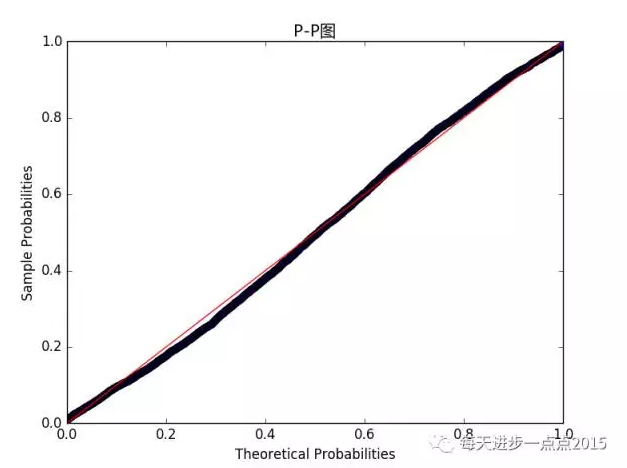
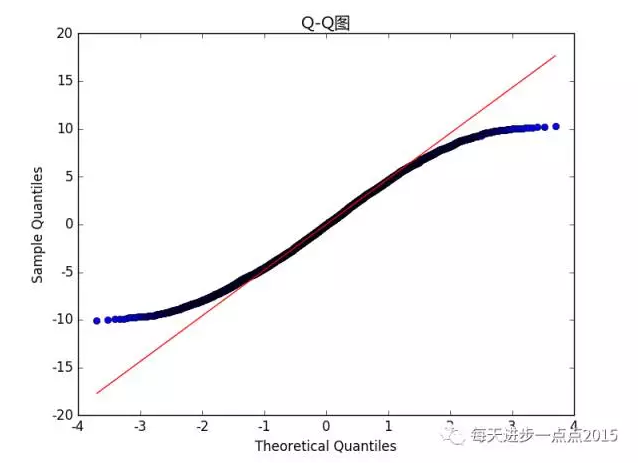
从PP图和QQ图来看,有一部分样本点并没有落在参考线上,但绝大多数样本点还是与参考线保持一致的步调。
# 残差的正态性检验(非参数法)
standard_resid = (resid-np.mean(resid))/np.std(resid)
stats.kstest(standard_resid, 'norm')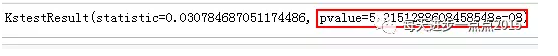
由于shapiro正态性检验对样本量的要求是5000以内;而本次数据集的样本量由9000多,故选择K-S来完成正态性检验。从K-S检验的P值来看,拒绝了残差服从正态分布的假设,即认为残差并不满足正态性假设这个前提。如果残差不服从正态分布的话,建议对Y变量进行box-cox变换处理。由于fit2模型的残差并没有特别明显的偏态(偏度为0.058,接近于0),故这里就不对Y变量进行box-cox变换了。如果需要变换的话,可以以下面的代码为例:
import scipy.stats as stats# 找到box-cox变换的lambda系数lamd = stats.boxcox_normmax(your_data_frame.y, method = 'mle')# 对Y进行变换your_data_frame['trans_y'] = stats.boxcox(your_data_frame.y, lamd)# 建模fit = sm.formula.ols('y~x1+x2+...', data = your_data_frame).fit()
fit.summary()关于线性回归模型的异常点识别、线性性假设、无多重共线性假设和残差的正态性假设在Python中的实现就介绍到这里。下一期,我们将针对线性回归模型残差的方差齐性和独立性假设进行讲解,接下来我们再用R语言对上面的内容复现一遍。
R语言脚本复现
# 加载第三方包
library(readxl)
library(GGally)
# 读取数据
ccpp <- read_excel(path = file.choose())
summary(ccpp)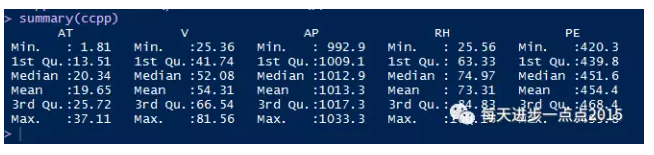
# 绘制各变量之间的散点图与相关系数
ggpairs(ccpp)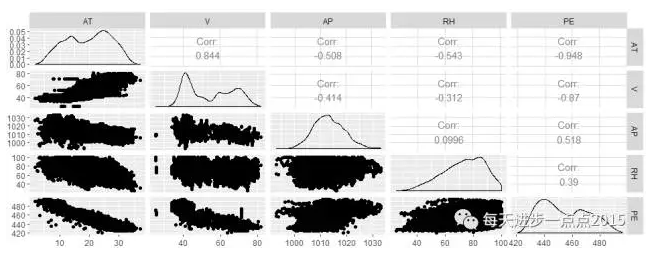
# 建模
fit <- lm(PE ~ AT + V + AP, data = ccpp)
summary(fit)
# 计算模型的RMSE值
RMSE = sqrt(mean(fit$residuals**2))
RMSE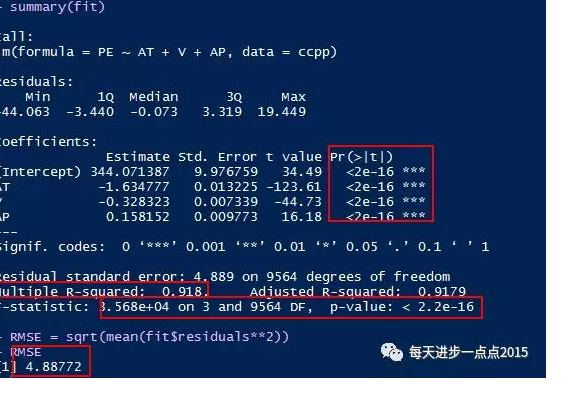
# 多重共线性检验
vif(fit)
# 异常点检验# 高杠杆值点(帽子矩阵)
leverage <- hatvalues(fit)
head(leverage)
# dffits值
Dffits <- dffits(fit)
head(Dffits)
# 学生化残差
resid_stu <- Dffits/sqrt(leverage/(1-leverage))
head(resid_stu)
# cook距离
cook <- cooks.distance(fit)
head(cook)
# covratio值
Covratio <- covratio(fit)
head(Covratio)
# 将上面的几种异常值检验统计量与原始数据集合并
ccpp_outliers <- cbind(ccpp, data.frame(leverage, Dffits, resid_stu, cook, Covratio))
head(ccpp_outliers)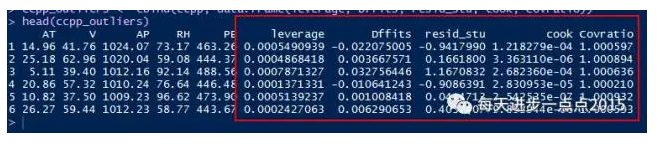
# 计算异常值数量的比例
outliers_ratio = sum(abs(ccpp_outliers$resid_stu)>2)/nrow(ccpp_outliers)
outliers_ratio
# 删除异常值
ccpp_outliers = ccpp_outliers[abs(ccpp_outliers$resid_stu)<=2,]# 重新建模
fit2 = lm(PE~AT+V+AP,data = ccpp_outliers)
summary(fit2)
# 计算模型的RMSE值
RMSE2 = sqrt(mean(fit2$residuals**2))
RMSE2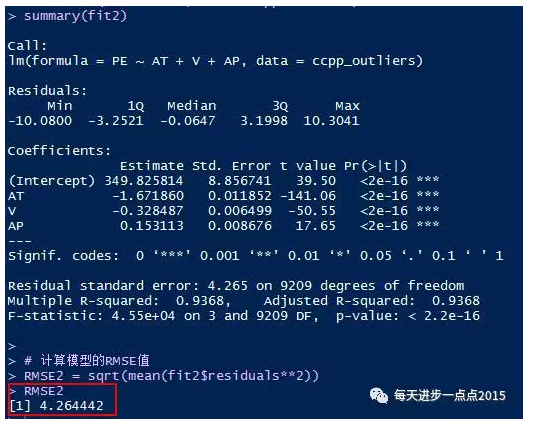
# 正态性检验#绘制直方图
hist(x = fit2$residuals, freq = FALSE,
breaks = 100, main = 'x的直方图',
ylab = '核密度值',xlab = NULL, col = 'steelblue')
#添加核密度图
lines(density(fit2$residuals), col = 'red', lty = 1, lwd = 2)
#添加正态分布图
x <- fit2$residuals[order(fit2$residuals)]
lines(x, dnorm(x, mean(x), sd(x)),
col = 'blue', lty = 2, lwd = 2.5)
#添加图例
legend('topright',legend = c('核密度曲线','正态分布曲线'),
col = c('red','blue'), lty = c(1,2),
lwd = c(2,2.5), bty = 'n')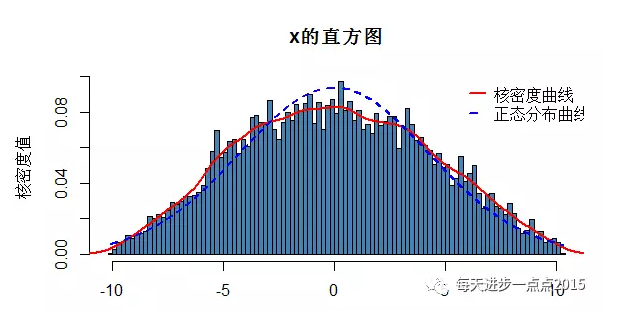
# PP图
real_dist <- ppoints(fit2$residuals)
theory_dist <- pnorm(fit2$residuals, mean = mean(fit2$residuals),
sd = sd(fit2$residuals))
# 绘图
plot(sort(theory_dist), real_dist, col = 'steelblue',
pch = 20, main = 'PP图', xlab = '理论正态分布累计概率',
ylab = '实际累计概率')
# 添加对角线作为参考线
abline(a = 0,b = 1, col = 'red', lwd = 2)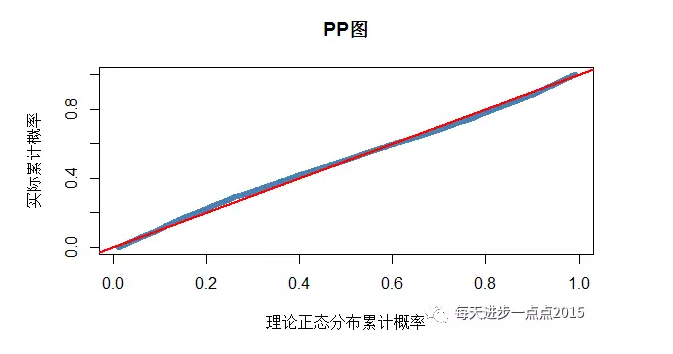
# QQ图
qqnorm(fit2$residuals, col = 'steelblue', pch = 20,
main = 'QQ图', xlab = '理论分位数',
ylab = '实际分位数')
# 绘制参考线
qqline(fit2$residuals, col = 'red', lwd = 2)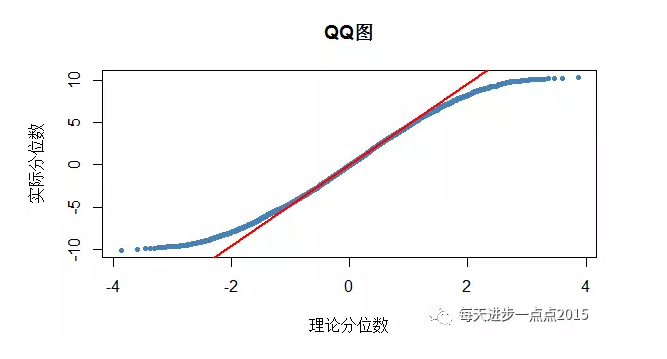
# shapiro正态性检验
# shapiro <- shapiro.test(fit2$residuals)
# shapiro
# K-S正态性检验
ks <- ks.test(fit2$residuals, 'pnorm',
mean = mean(fit2$residuals),
sd = sd(fit2$residuals))
ks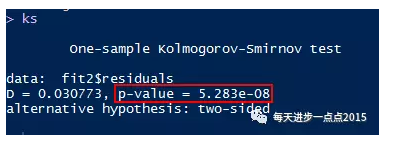
结语
OK,今天关于线性回归诊断部分的内容就分享到这里,限于篇幅,不能一次性写完,故在下一期将继续推出诊断的其他内容(残差的方差齐性和残差的独立性检验)。希望对数据挖掘或机器学习比较感兴趣的朋友,能够静下心来好好的复现一遍。如果你有任何问题,欢迎在公众号的留言区域表达你的疑问。同时,也欢迎各位朋友继续转发与分享文中的内容,让更多的朋友学习和进步。
关注“每天进步一点点2015”
相关材料下载链接
链接: https://pan.baidu.com/s/1qYNsP0w 密码: 2g3f